如何分析GEO数据库中某一疾病的差异基因?
1、一、查找数据(以骨肉瘤数据为例)
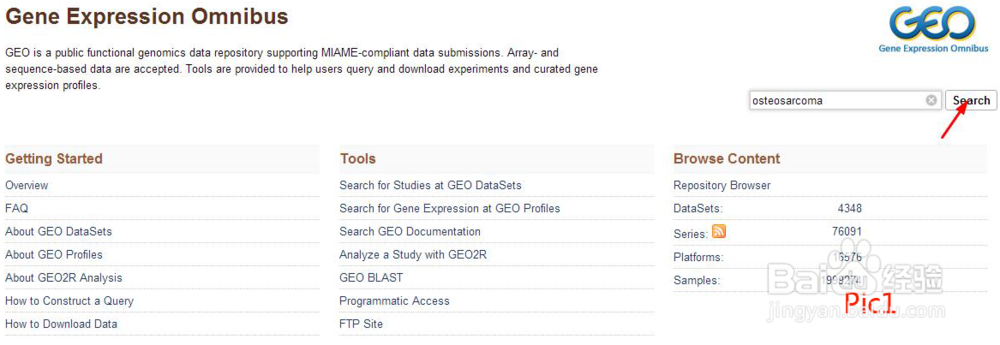
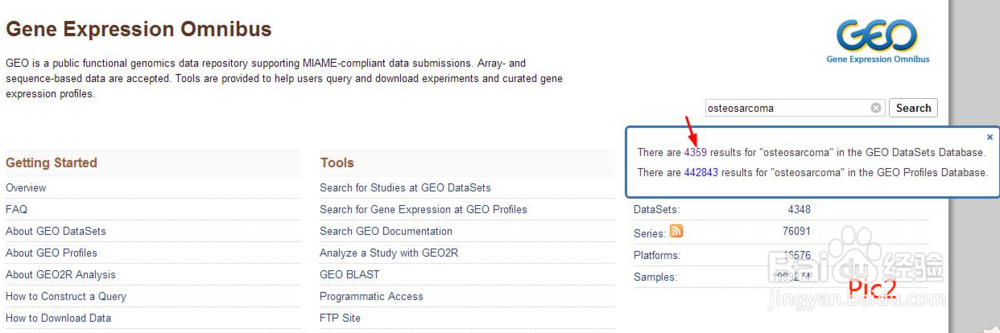
1、打开GEO数据库,如pic1所示,输入osteosarcoma搜索。
2、出现pic2所示对话框,点击“There are 4359 results for "osteosarcoma" in the GEO DataSets Database”数字处进入。
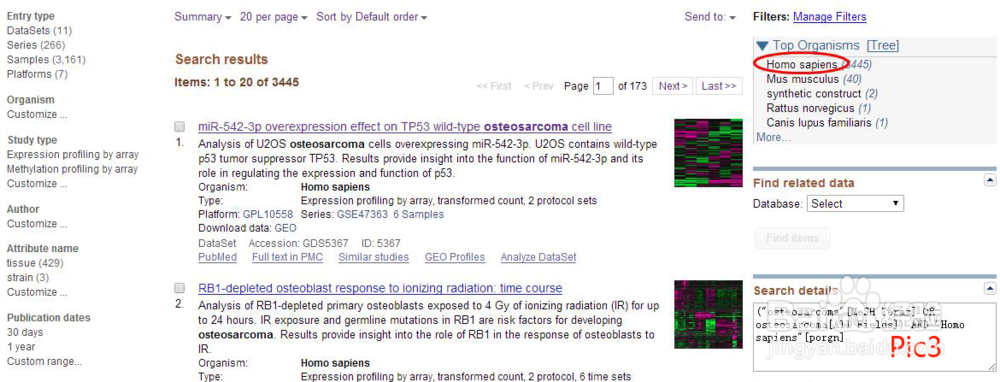
3、物种选择“Homo sapiens” ,如pic3所示,点击选择。
4、数据选择
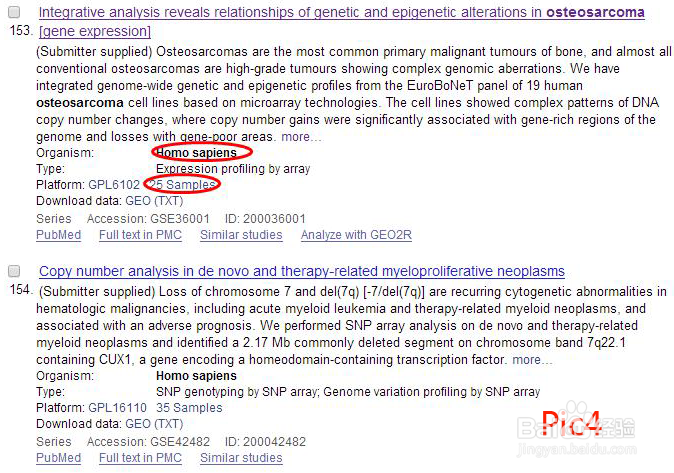
查看题目及简介,确定是否为所需内容。然后查看“Samples”的数量是否合适(大于20且小于1000为宜),“Type”是否为芯片数据,如pic4。
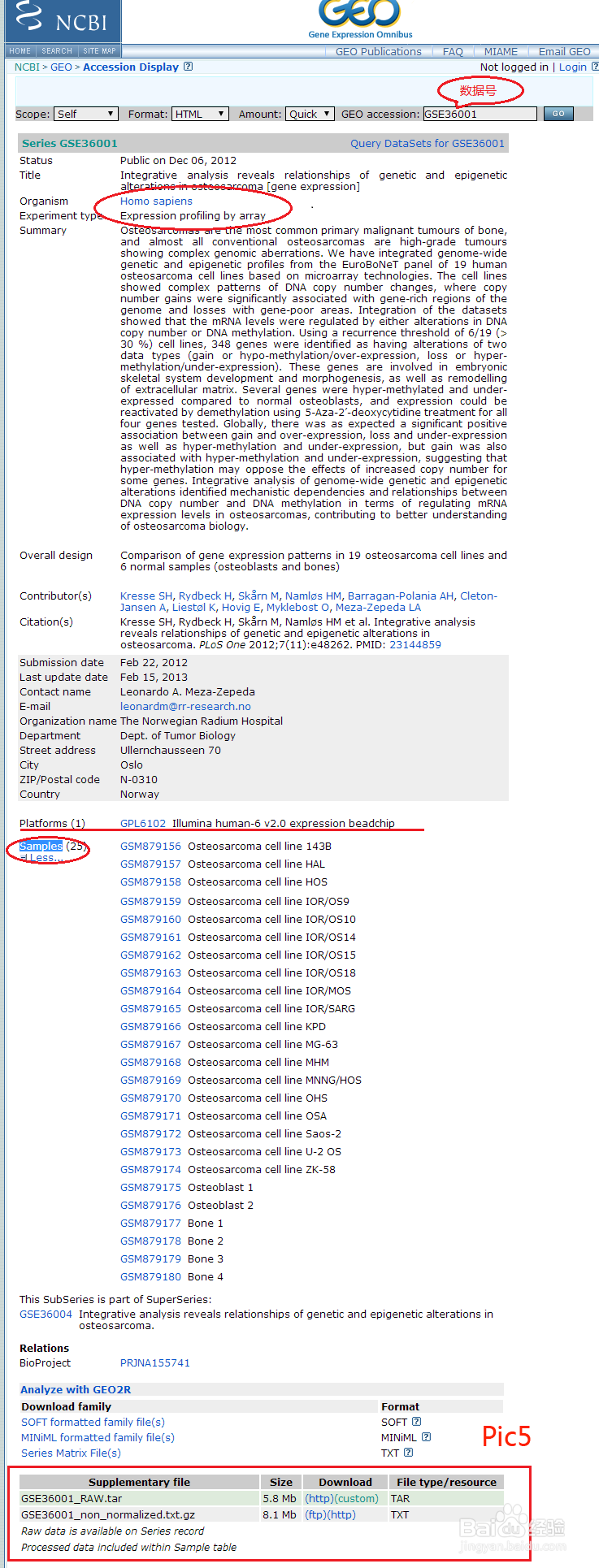
5、点击进入,查看其中内容,如pic5:
①Platforms (平台)是否可用(可用平台为:illuminaHumanv1,illuminaHumanv2,illuminaHumanv3,illuminaHumanv4,hgu133a,hgu133a2,hgu133b,hgu95a,hgu95av2, hgu133plus2, hgu219,hugene10st, InfiniumMethylation)。
②查看有无 “Processed data”(必有) 及 “Raw data”(可有可无),
③点开“Samples”处确认有对照组,本例中为“Bone ”与“Osteosarcoma cell line”为对照,
④满足以上条件,记录数据号备用。
1、二、本地化处理
1、搜索平台 基因云馆,登录系统(或注册后登录),如pic6。
2、在左侧模块栏选择“数据库”(pic7)中的GEO数据库(pic8),在queryNAME*处输入对应的数据号“GSE36001”,type处选择“处理后的数据”,点击“运行”。

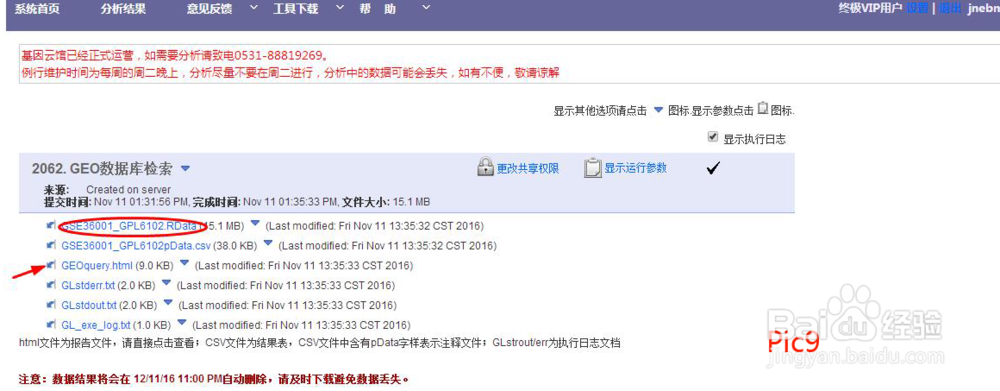
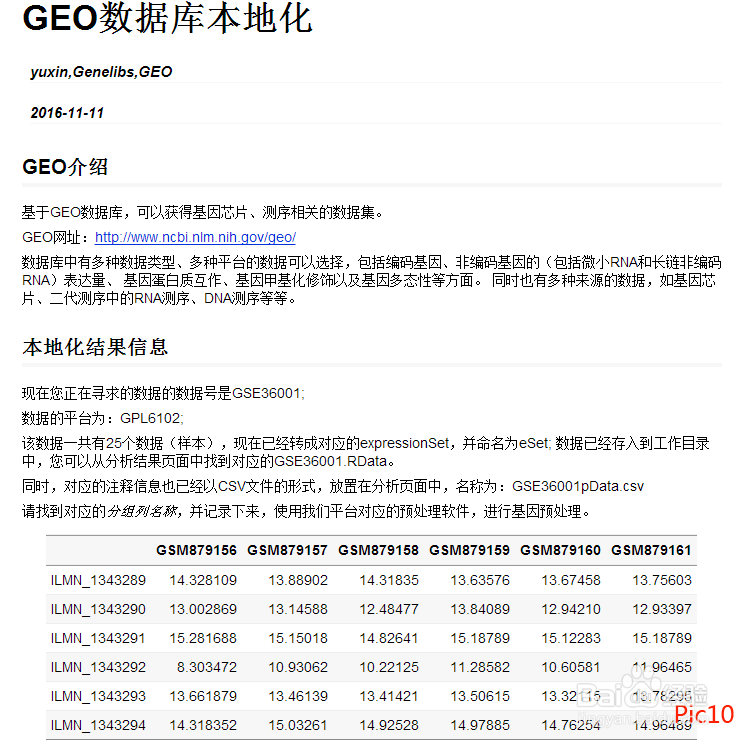
3、运行结束,生成RData文件(pic9)及报告等内容。点击html文件查看报告(pic10):列名称有无空格,若有则参照预处理步骤进行更改。
1、三、预处理
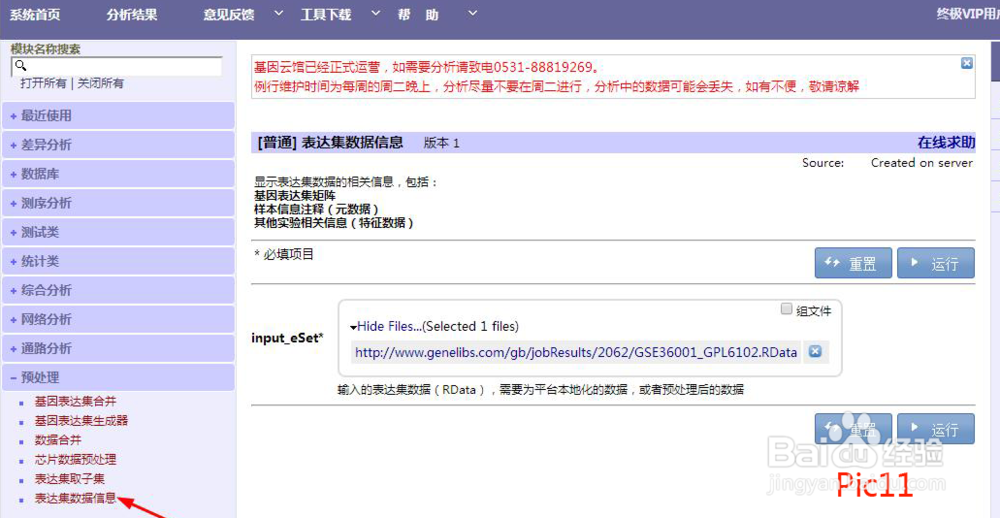
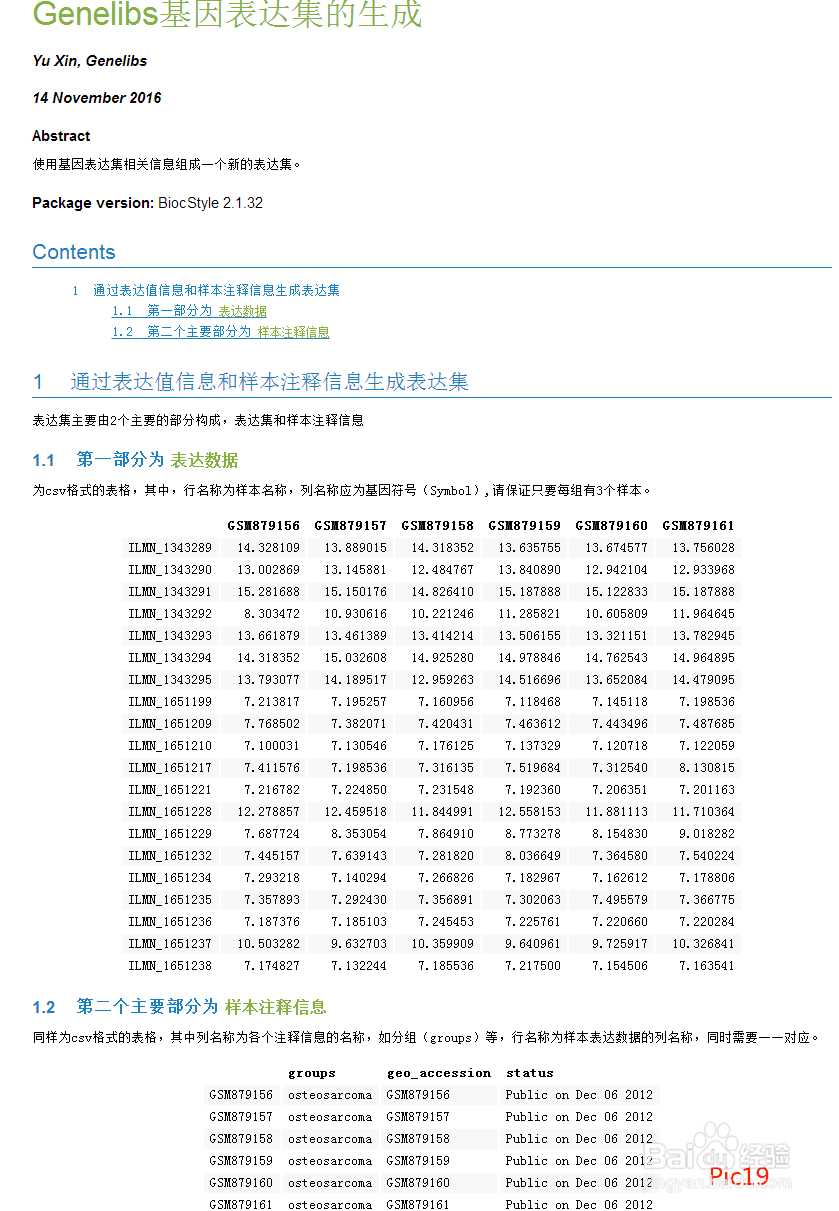
1、表达集数据信息(此步骤在数据信息需要更改时配合“基因表达集生成器”使用,以确保修改后PData的行名称和RData的列名称保持一致),
在预处理模块选择“表达集数据信息”,将本地化生成的RData文件放入input_eSet中,点击“运行”(pic11),
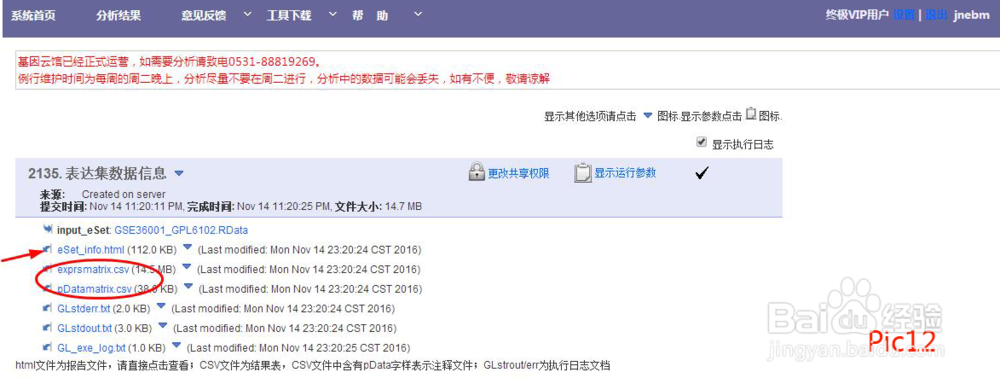
运行结束会生成两个“csv”文件及报告等(pic12)。点击html文件查看报告(pic13),
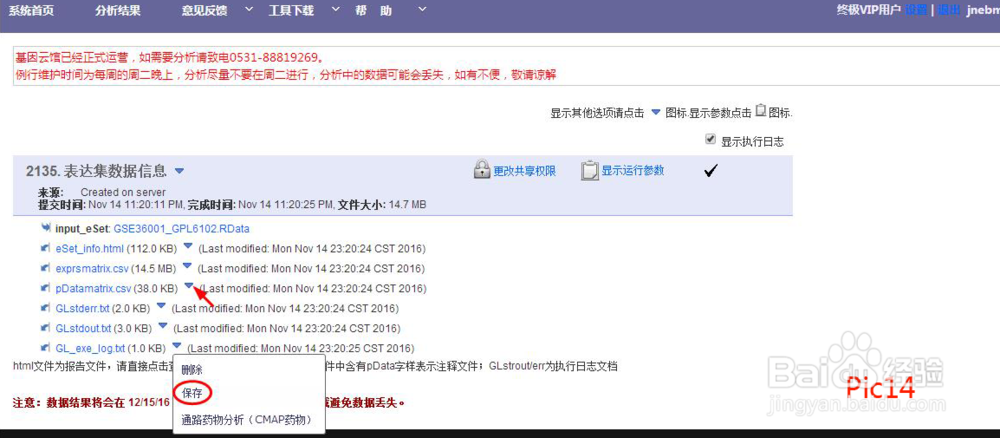
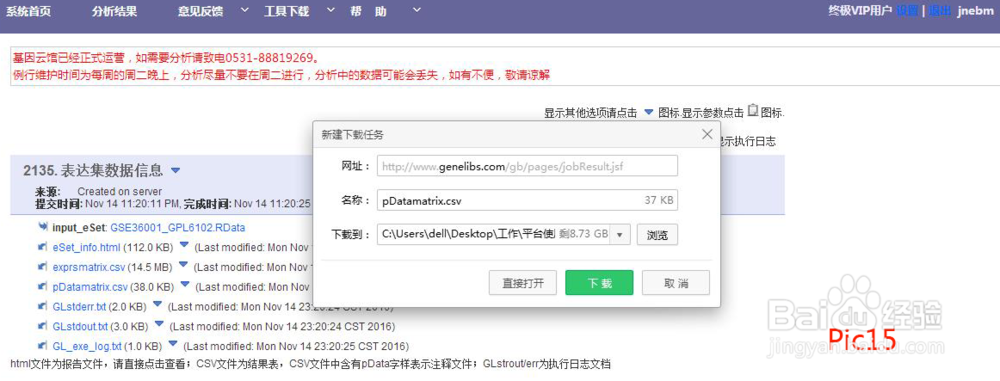
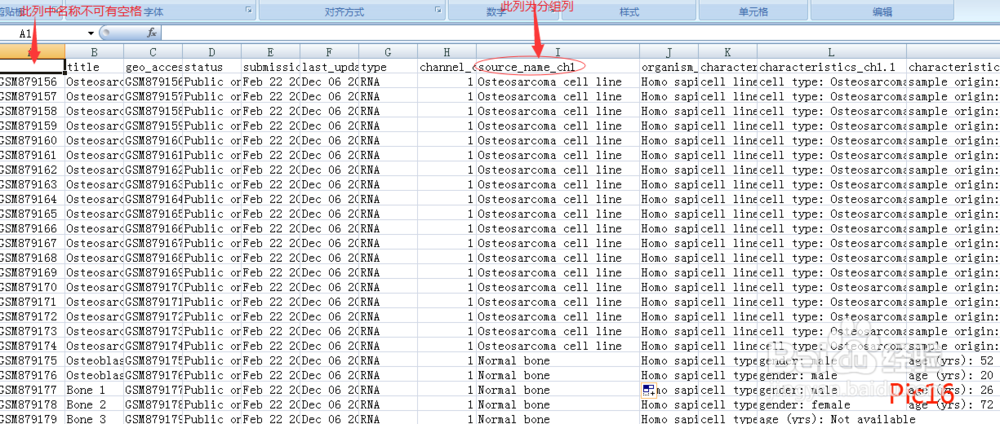
将生成的“pDatamatrix.csv”保存(如pic14、pic15所示 )后根据需要进行更改(第一个csv文件的列名称与第二个csv文件的行名称对应且无空格;分组列中名称后无空格且分组名称尽量简单)(如pic16),修改完成后保存备用。(如PData的行名称更改,则exprsmatrix.csv的列名称也要做相应的改变)。

2、基因表达集生成器
在预处理模块选择“基因表达集生成器”,将修改好的“exprsmatrix.csv”文件放入matrix*;“pDatamatrix.csv ”文件放入pData*中,根据需要填写保存名称,运行即可,如pic17所示,
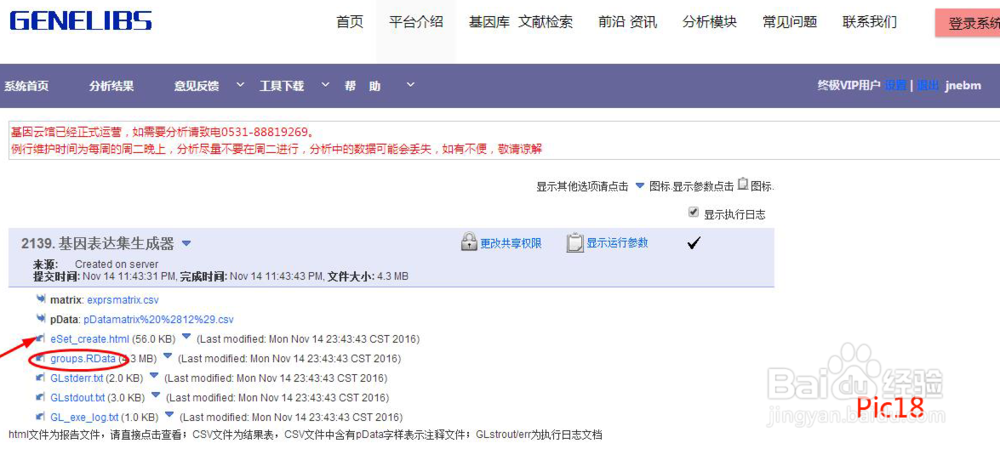
运行结束生成一个新的RData文件及报告等(如pic18所示)。点击html文件查看报告(如pic19所示)。

3、芯片数据预处理
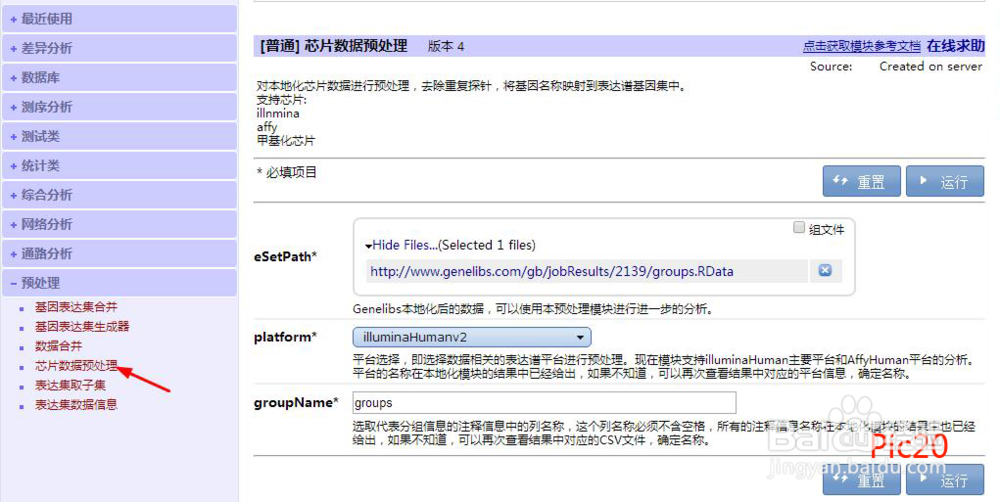
在预处理模块选择“芯片数据预处理”,将新生成的RData文件放入eSetPath*,在platform*处选择相应平台(pic5中已给出),groupName*处填入分组列的列名称,点击运行,如pic20,
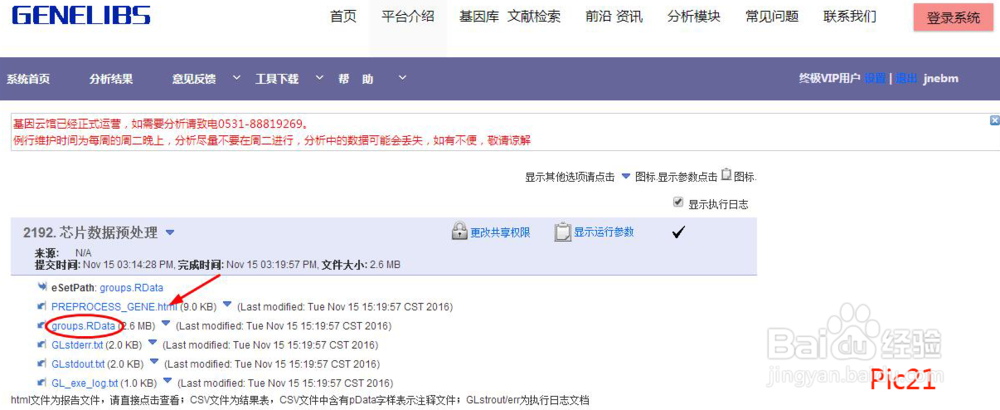
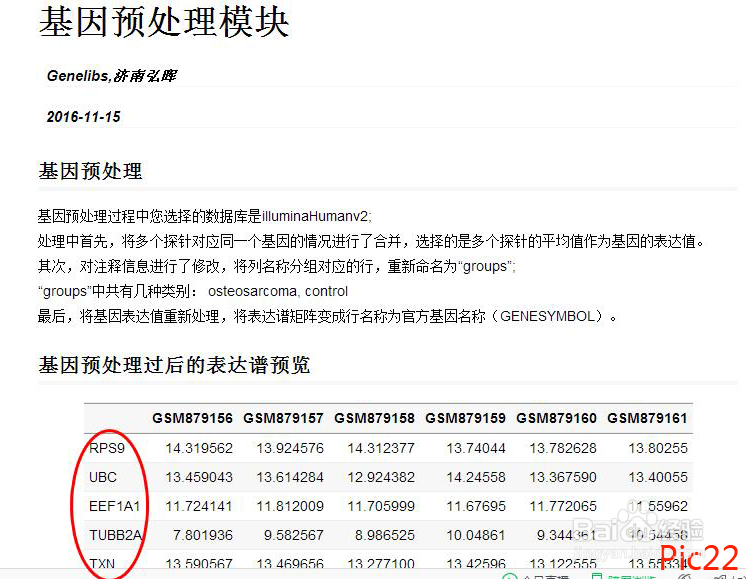
运行结束会生成新的RData文件及报告(如pic21),点击html文件查看报告(如pic22),此时表达谱列名称已转换为基因名称。
1、四、差异基因分析
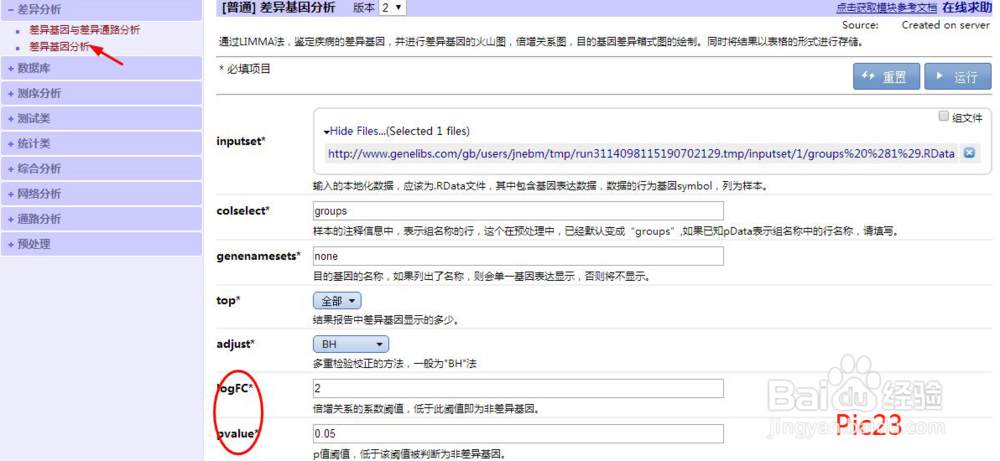
在inputset*处放入新生成的RData文件,colselect*处填写分组信息的列名称,根据需要更改阈值,点击运行(pic23),
运行结束,生成html报告等内容(pic24),
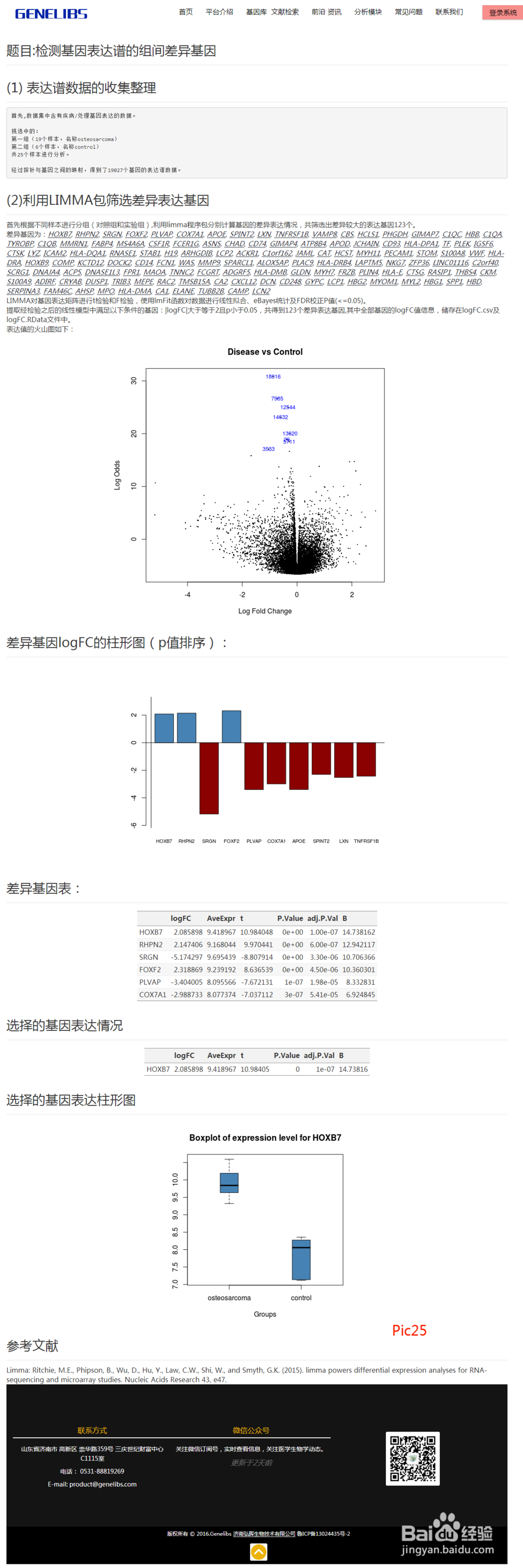
点击html查看报告如pic25,可知差异基因等,
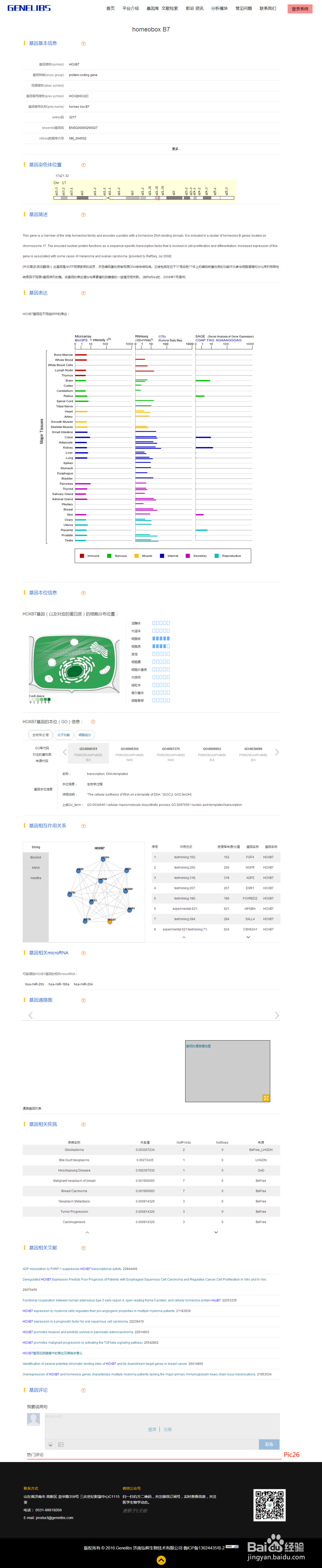
点击报告中某一差异基因,可查看该基因的具体信息,如pic26。