用python程序输出DNA互补序列,碱基数
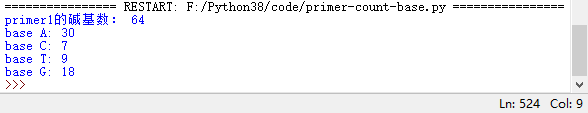
1、先说简单的,统计DNA长度,碱基数量。输入DNA序列,记得要加引号
primer1='GACAAAAAAAAAAAAAAAAAAAATGTAGAGTAGGGACGCACAGAGGCTGAGTCGTTGTGATCGG'

2、输出引物碱基总数,在python里就是用len()函数计算字符串长度
print('primer1的碱基数:',len(primer1))
3、输出各个碱基的数量,这里使用了一个 .count('')方法,效果是统计字符串内某字符的数量。
print('base A:',primer1.count('A'))
print('base C:',primer1.count('C'))
print('base T:',primer1.count('T'))
print('base G:',primer1.count('G'))
4、互补序列的计算。使用 .translate()方法获得互补序列,括号内是采用的翻译列表;使用str.maketrans('','')方法建立翻译列表,这里就是互补规则:AT,CG互补
table=str.maketrans('ACGT','TGCA')#碱基互补翻译表
print(table)
Baserc=primer1.translate(table)

5、注意,这里的序列碱基虽然是互补的,但是方向是同向的,双链DNA是反向互补,所以必须将序列反向。我不知道如何将字符串反向,所以新建了一个序列,反向填充Baserc的字符,然后把序列转化成字符串。
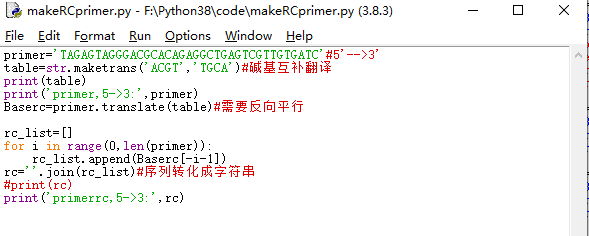
6、输出序列及其互补序列
print('primer1,5->3:',primer1)
print('primer1rc,5->3:',rc)
声明:本网站引用、摘录或转载内容仅供网站访问者交流或参考,不代表本站立场,如存在版权或非法内容,请联系站长删除,联系邮箱:site.kefu@qq.com。
阅读量:156
阅读量:84
阅读量:119
阅读量:88
阅读量:68